Elaboración de mapa de NDVI con tmap
Sentinel-2 es una misión de imágenes multiespectrales de alta resolución que apoya los estudios de monitoreo de tierras de Copérnico, incluyendo el monitoreo de la vegetación, el suelo y la cubierta del agua, así como la observación de vías navegables interiores y áreas costeras.
Y una de las formas de procesar estos datos satelitales es Google Earth Engine (GEE), una plataforma basada en la nube para el análisis geoespacial a escala planetaria (Gorelick et al., 2017).
En el presente ejemplo se presentará algunos pasos que permite la generación de mapas usando directamente los datos del dataset Sentinel-2 MSI: MultiSpectral Instrument, Level-2A de GEE mediante el lenguaje R y algunos de sus paquetes que permiten tal proceso.
Para comenzar necesitará tener los archivos del área de interés descagar y ubicarlos en una carpeta dentro de su proyecto. Y se usará los paquetes rgee, sf, raster, tidyverse y tmap.
Librerías necesarias
Leer la documentación en github para su adecuada instalación de rgee. Los demás paquetes si se puede instalar normalmente con la función pacman::p_load() si es que no lo tiene con anterioridad.
## Carga de librerías
require(pacman)
pacman::p_load(
rgee, sf, raster, tidyverse, tmap
)Inicio de sesión de Google Earth Engine
## Iniciar la sesión de GEE
rgee::ee_Initialize()Filtro de nube
El dataset Sentinel-2 MSI: MultiSpectral Instrument, Level-2A ya se encuentran procesadas con sen2cor. Solo sería necesario filtrar las nubes.
## Código de filtro de nubes
## https://csaybar.github.io/blog/2020/06/15/rgee_02_io/
getQABits <- function(image, qa) {
# Convert decimal (character) to decimal (little endian)
qa <- sum(2^(which(rev(unlist(strsplit(as.character(qa), "")) == 1))-1))
# Return a single band image of the extracted QA bits, giving the qa value.
image$bitwiseAnd(qa)$lt(1)
}
s2_clean <- function(img) {
# Select only band of interest, for instance, B2,B3,B4,B8
img_band_selected <- img$select("B[2-4|8]")
# quality band
ndvi_qa <- img$select("QA60")
# Select pixels to mask
quality_mask <- getQABits(ndvi_qa, "110000000000")
# Mask pixels with value zero.
img_band_selected$updateMask(quality_mask)
}Variables en GEE
## Área de interés
box <- ee$Geometry$Rectangle(coords = c(-76.97,-12.17,-76.90,-12.10),
## WGS 84
proj = "EPSG:4326",
geodesic = FALSE
)
## Filtrar las nuebes de la imagen seleccionada
image <- ee$ImageCollection(ee$Image("COPERNICUS/S2_SR/20191028T151711_20191028T152253_T18LTM"))$
map(s2_clean)$first()
## Generar imagen NDVI
ndvi <- image$normalizedDifference(c("B8", "B4"))Área de la Loma
Cargar el archivo shape descargado.
## Dirección de los archivos shape descargados
datos_mapa <- "dirección del shape" ## Ejemplo: "data/ACR/ACR.shp"
## Shaoe del área de interés
loma_shape <-
## Cambiar la ubicación en donde usted guardó su archivo shapefile
raster::shapefile(datos_mapa) %>%
## Convertir sp a sf
sf::st_as_sf(loma_shape) %>%
## Obtener el shape de Lomas de Villa María
dplyr::filter(acr_codi == "ACR23" & objectid == 2924) %>%
## UTM 18S
sf::st_transform(
crs = "+proj=utm +zone=18 +south +ellps=WGS84 +datum=WGS84 +units=m +no_defs"
)Obtención del raster de NDVI
Guardar en una variable la imagen raster procesada en GEE.
## Raster de NDVI del área de interés
raster_ndvi <-
## Convertir objeto ee a raster
rgee::ee_as_raster(
image = ndvi,
region = box,
scale = 10
) %>%
## UTM 18S
raster::projectRaster(
crs = "+proj=utm +zone=18 +south +ellps=WGS84 +datum=WGS84 +units=m +no_defs"
) %>%
raster::crop(loma_shape) %>%
raster::mask(loma_shape)Mapa con tmap
Guardar en una variable el mapa generado.
map_ndvi <- tm_shape(raster_ndvi) +
## Características del raster en el mapa
tm_raster(style = "fixed", title = "NDVI",
palette = c("#ff0707","#fff823","#45ff17","#13deff",
"#1d4eff","#dc1dff","#be3ea8"),
legend.hist = TRUE, n = 6,
breaks = c(raster::minValue(raster_ndvi), 0.1, 0.2, 0.3, 0.4,
0.5, 0.6, raster::maxValue(raster_ndvi))) +
tm_legend(outside = TRUE) +
## Añadir un barra de escala al mapa
tm_scale_bar(
position = c("left", "bottom"), breaks = c(0, 0.5, 1),
text.size = 0.7
) +
tm_compass(
size = 2, position = c("right", "top"),
type = "4star"
) +
## Añadir los créditos del mapa
tm_credits(text = "Poner su nombre",
size = 0.8, position = c("right", "bottom")) +
## Uso de un estilo predeterminado
tm_style("cobalt", legend.position = c("left","center"),
legend.format = list(text.separator= "-")) +
## Arreglos generales del mapa a generar
tm_layout(
title = "Título del mapa",
title.position = c("center", "top"),
inner.margins = 0.05
)
map_ndviGuardar el mapa
Puede guardar en diferentes formatos los mapas generados.
## png
tmap::tmap_save(
tm = map_ndvi,
filename = "Nombre de archivo de salida", ## Ejemplo: "map_ndvi.png"
units = "cm", height = 15, width = 15,
dpi = 900
)
## pdf
tmap::tmap_save(
tm = map_ndvi,
filename = "Nombre de archivo de salida", ## Ejemplo: "map_ndvi.pdf"
units = "cm", height = 15, width = 15,
dpi = 900
)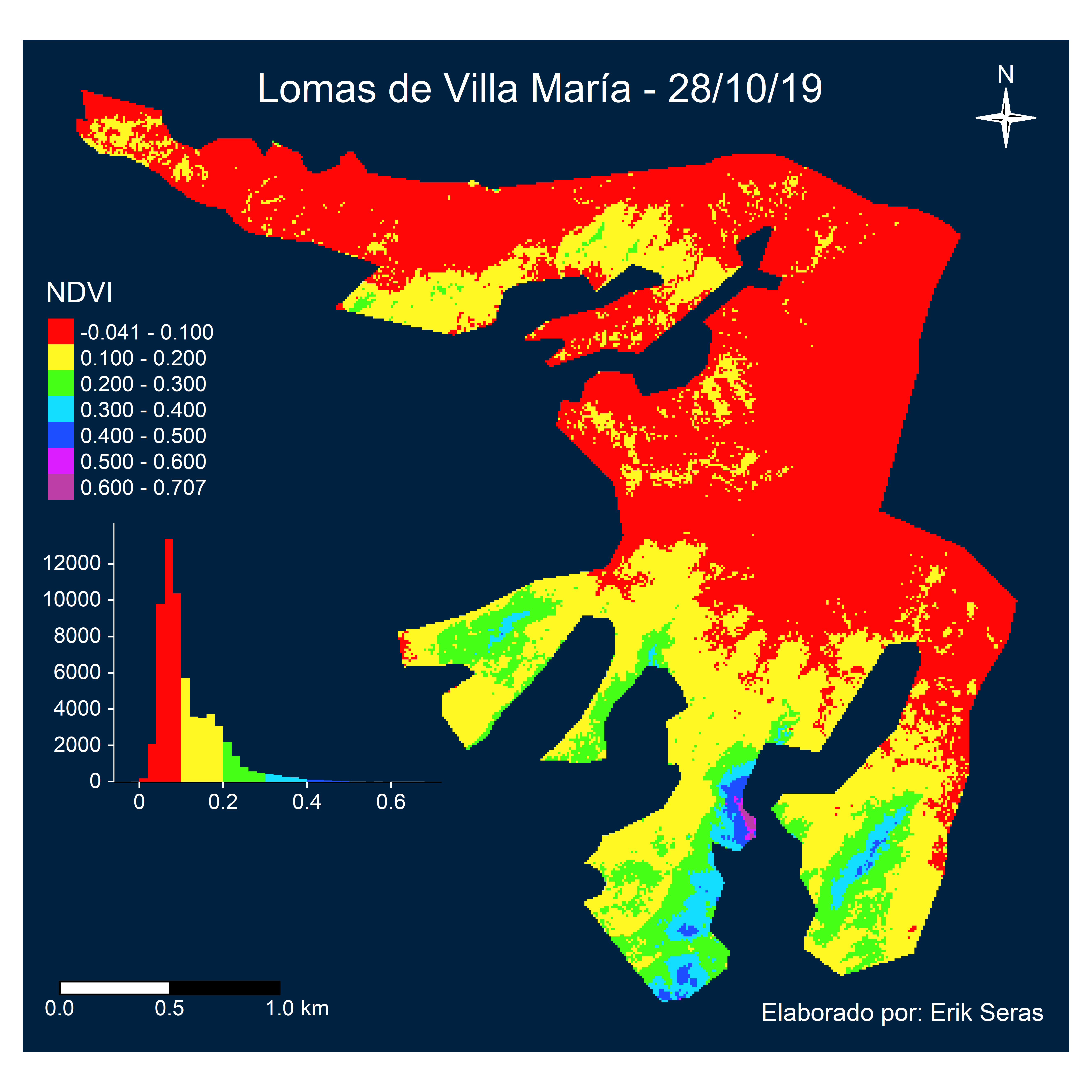
Ejemplo de mapa generado en formato png
